Was ist Wirkstoffdesign?
Eine Einführung für Nicht-Fachleute
Arzneimittel hat vermutlich schon jeder von uns eingenommen. Vielleicht hat sich der eine oder andere dabei gefragt, was passiert eigentlich mit meinem Körper, wenn sich die Tablette auflöst und die darin enthaltenen Stoffe freigesetzt werden. Wieso findet das Aspirin den Kopfschmerz und kann mich von dieser Pein befreien?
Nach der Einnahme beginnt sich eine Tablette im Magen oder Darm aufzulösen und setzt den darin enthaltenen Arzneistoff, in aller Regel eine niedermolekulare organische Verbindung, frei. Diese überwinden dann die Magen- und Darmwand und gelangen in die Blutbahn. Von dort beginnt der Transport durch den Körper, der zunächst über die Leber führt. Ziel dieses Transportes ist der Ort, an dem ein Arzneistoff seine Wirkung entfalten soll. Diese Orte sind meist Rezeptoren oder biochemische Katalysatoren (Enzyme), die wichtige Funktionen in unserem Körper ausüben. Beispielsweise befinden sich Rezeptoren an der Oberfläche von Zellen, und durch Bindung eines kleinen Moleküls wird der Zelle ein Signal vermittelt, so dass im Inneren der Zelle Prozesse in Gang gesetzt werden. Der Körper produziert selbst solche Signalstoffe, wie zum Beispiel das Adrenalin. Arzneistoffe können nun mit diesen Botenstoffen um die Rezeptoren konkurrieren und so die Aufgaben einer Zelle beeinflussen, in dem Prozesse entweder gezielt ausgelöst oder blockiert werden. Die genannten biochemischen Katalysatoren oder Enzyme sind für Umwandlungen von Stoffen in unserem Körper verantwortlich. Mit ihrer Hilfe werden die Bausteine synthetisiert und chemisch umgewandelt, die wir für alle Vorgänge des Lebens brauchen; angefangen von der Verstoffwechselung unserer Nahrung bis hin zu Prozessen, die bei einer blutenden Wunde dafür sorgen, dass die Wunde wieder verschlossen wird.
Auch der Inhaltsstoff des Aspirins, Acetylsalicylsäure, greift an einem solchen Enzym an und beeinflusst dessen Funktion. Es spielt in der Biosynthese körpereigener Verbindungen (so genannter Entzündungsmediatoren) eine wichtige Rolle, die vermehrt in entzündetem Gewebe gebildet werden und unter anderem die Schmerzrezeptoren sensibilisieren. Aspirin vermag die Bildung dieser Verbindungen herunter zu regeln.
Damit nun ein Arzneistoff die Funktion eines Enzyms beeinflussen kann, muss es spezifisch und selektiv an diesen Katalysator binden. Die Stelle, an der normalerweise die Umwandlung der körpereigenen Stoffe durchgeführt wird, muss blockiert werden. Man kann diese Aufgabe mit der Funktion eines Schlüssels in einem Schlossvergleichen (Abb. 1 links). Nur wenn ein Schlüssel die richtige Form und Gestalt besitzt, passt er in das Schloss und kann seine Funktion ausüben. Arzneistoffe, die ihre Wirkung als Enzyminhibitoren entfalten, haben zwar die richtige Passform des Schlüssels, aber sie setzen die Funktion des Schlosses außer Kraft. Man kann dies mit dem Abbrechen des Schlüssels im Schloss vergleichen.
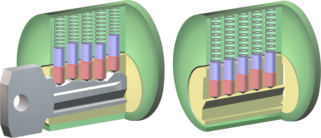
Abb. 1
Der Arzneimittelforscher versucht nun optimale Schlüssel für ein vorgegebenes Schloss zu finden. Diese Suche nach richtigen Schlüsseln für das Schloss ist Thema unserer Forschung. Was ist dazu nötig? Zum einen muss man die dreidimensionale Geometrie des Schlosses kennen. Die Raumstruktur von Enzymen und Rezeptoren lässt sich mit der Röntgenstrukturanalyse aufklären. Dazu müssen diese sehr großen Biomoleküle kristallisiert werden. In einem Kristall finden sich sehr viele dieser Moleküle zusammen und ordnen sich regelmäßig nebeneinander an. Kristalle sind wiederum in der Lage, Röntgenstrahlen zu beugen. Aus dem erzeugten Beugungsbild kann man dann zurückrechnen, welchen strukturellen Aufbau die Moleküle besitzen, die in dem Kristall das Beugungsbild erzeugt haben.
Ein solches computergesteuertes Gerät, mit dem man Röntgenbeugungsexperimente an Kristallen durchführen kann, haben wir bei uns im Institut aufgebaut oder wir gehen mit unseren Kristallen an ein Synchrotron und bestimmen damit die Strukturen unserer Schlösser. Damit kennen wir natürlich noch nicht vermeintlich passende Schlüssel. Ist allerdings ein solcher Schlüssel bekannt, so kann man seine Bindungsgeometrie, sprich wie er sich im Schloss anordnet, einfach bestimmen, indem man erneut die Kristallstruktur des Schlosses, jetzt aber mit gebundenem Schlüssel, bestimmt (Abb. 2).
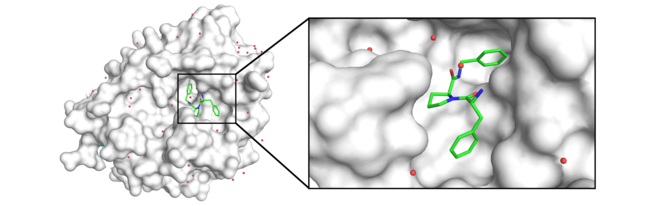
Abb. 2
Die Suche nach neuen Schlüsseln ist der Schwerpunkt unserer Arbeitsgruppe. Man kann einerseits von der Gestalt der Bindetasche des Enzyms oder Rezeptors ausgehen und sich anhand dieser Vorlage neue Schlüssel zurechtfeilen (Abb. 2 rechts). Andererseits kann man sich große Sammlungen von Schlüsseln, sprich Datenbanken mit Molekülen, vornehmen und einen Schlüssel nach dem anderen durchprobieren, ob er in das vorgegebene Schloss passt. Diese aufwändige Analyse kann nur mit dem Computer durchgeführt werden; daher entwickeln wir Docking-Programme, die uns bei dieser Aufgabe unterstützen. Diese Suchverfahren setzen voraus, dass man verstanden hat, welche Kriterien dafür verantwortlich sind, so dass ein Schlüssel tatsächlich für ein gegebenes Schloss geeignet ist. So schön der Vergleich von Schlüssel und Schloss sein mag, er vereinfacht die bei wechselwirkenden Molekülen gegebenen Verhältnisse. Moleküle sind konformativ beweglich, sie können unterschiedliche Arten von Wechselwirkungen aufbauen. All diese Aspekte müssen richtig verstanden sein, damit die Suche erfolgreich ist. Leider sind wir erst dabei, die Kriterien zusammenzustellen und sie langsam zu verstehen. Wir führen deshalb auch Experimente durch, die uns Aufschluss über die Kriterien geben, die letztlich bestimmen, warum ein Arzneistoff an ein bestimmtes Protein bindet. Gleichzeitig müssen wir uns neue Moleküle herstellen, die als Ideen aus unseren Computersuchen entstanden sind. Diese Arbeiten führen wir häufig auch mit Kollegen durch, die die Synthese neuer Verbindungen als Schwerpunkt ihrer Arbeiten haben. Wenn wir verstehen wollen, warum ein bestimmter Schlüssel passt, versuchen wir bei unserer Schlüsselsuche das Pferd auch von hinten aufzuzäumen. Wir können nicht nur unsere Schlüssel strukturell abwandeln, wir können dies auch mit unseren Schlössern durchführen. Die Bindetaschen in Enzymen und Rezeptoren sind aus Aminosäuren zusammengesetzt. Diese Aminosäuren lassen sich austauschen und man kann verfolgen, wie sich diese Änderung auf die Bindung eines Arzneistoffs auswirkt. Gleichzeitig lernt man mehr über die Funktion eines Biomoleküls. Wir nehmen die Austausche (Mutationen) gezielt vor. Auch die Natur bedient sich vielfach dieser Veränderungen (Evolution). Beispielsweise unterscheiden sich funktionsgleiche Enzyme und Rezeptoren aus dem Menschen, aus unterschiedlichen Tieren oder Bakterien durch solche Austausche von Aminosäuren. Weiterhin werden Resistenzen gegen bestimmte Arzneistoffe genau durch solche Austausche erzeugt. Für unsere Schlüsselsuche ist es sehr wichtig zu verstehen, auf welchen Kriterien die Selektivitätsunterschiede zwischen ähnlichen Schlössern beruhen bzw. wie es zu Resistenzen gegen potente Arzneistoffe kommt. Man will beispielsweise mit einem Arzneistoff die Enzyme eines Bakteriums oder Virus treffen, die des Menschen müssen dabei aber unberührt bleiben. Hier hilft kein Generalschlüssel, hier muss ein selektiver Spezialschlüssel bereitstehen.
Nicht von jedem Schloss gelingt es, die Struktur aufzuklären. Dennoch braucht man auch in diesen Fällen Schlüssel als potentielle Arzneistoffe. Man kann einerseits versuchen, durch den Vergleich bereits bekannter Schlüssel, die vielleicht in mancher Hinsicht nicht optimal sind, neue Schlüssel zu finden. Auch die Strukturen verschiedener Schlüssel, die alle in ein unbekanntes, aber gemeinsames Schloss passen, enthalten viel Information über dieses Schloss (Abb. 3). Diese Information gilt es herauszulesen und auszunutzen.

Abb. 3
Andererseits kann man auch versuchen, ein Modell des unbekannten Schlosses zu bauen. Modelle lassen sich nur entwerfen, wenn man gewisse Vorstellungen hat, wie das Schloss in etwa auszusehen hat, sprich, es müssen verwandte Schlösser bekannt sein. Darüber kann man sich nur klar werden, wenn man die vorhandenen Daten sichten kann. Immerhin sind heute bereits die Kristallstrukturen von ca. 170.000 Proteinen aufgeklärt. Aus diesem Grund wird in unserer Arbeitsgruppe eine Datenbank entwickelt, in der man sowohl nach Strukturen der Enzyme und Rezeptoren suchen kann, aber auch gleichzeitig Fragen an kleine Moleküle richten kann, die an diese Proteine gebunden sind. Durch Auswertung dieser Daten wollen wir herausfinden, ob es Gemeinsamkeiten in der Gestalt der Bindetaschen gibt, die kleine Moleküle, z.B. Arzneistoffe, aufnehmen. Wir hoffen, dass wir anschließend diese Erkenntnisse für die Suche und den gezielten Entwurf neuer Arzneistoffe ausnutzen können.
Abbildungen:
Abb1: CC BY-SA 3.0, abgerufen am 24.01.2017, 12:00 Uhr
https://de.wikipedia.org/wiki/Schlie%C3%9Fzylinder#/media/File:Pin_tumbler_no_key.svg
https://de.wikipedia.org/wiki/Schlie%C3%9Fzylinder#/media/File:Pin_tumbler_with_key.svg
Abb2: Kristallstruktur Thrombin (PDB: 2ZFF)
Baum, B., Mohamed, M., Zayed, M., Gerlach, C., Heine, A., Hangauer, D., & Klebe, G. (2009). More than a Simple Lipophilic Contact: A Detailed Thermodynamic Analysis of Nonbasic Residues in the S1 Pocket of Thrombin. Journal of Molecular Biology, 390, 56–69. http://doi.org/10.1016/j.jmb.2009.04.051
Abbildung von Tobias Wulsdorf
Abb3: CC 1.0, abgerufen am 24.01.2017, 12:00 Uhr
http://www.publicdomainpictures.net/view-image.php?image=8178&picture=rusty- keys
Seitenanfang